Responsable du thème: Gaël Yvert
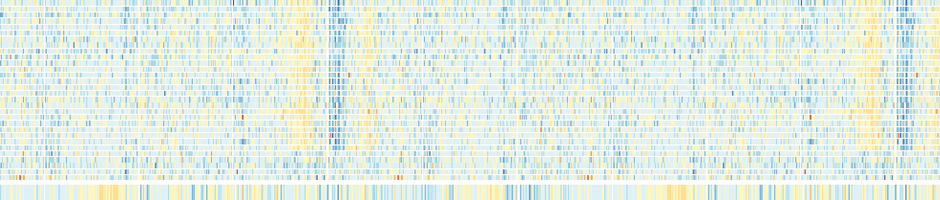
La chromatine porte de nombreuses informations qui ne sont pas écrites dans la séquence ADN elle-même. Le support moléculaire de cette information est assuré par des modifications chimiques des constituants de la chromatine, comme l'ajout d'un groupement méthyl sur les cytosines de l'ADN ou sur certaines lysines des protéines histones, ou encore l'ajout ou le retrait de groupement acetyl ou d'ubiquitine sur certains résidus des histones, ou même le remplacement d'une histone par une protéine très similaire mais différente: un variant d'histone. La séquence de cette information qui peut être lue le long des chromosomes est communément appelée l'épigénome.
Nous cherchons à déterminer le degré de variabilité naturelle inter-individus de l'épigénome. Pour un type cellulaire donné, à un âge donné et dans un environnement donné, deux individus portent potentiellement des épigénomes différents, et ces différentes peuvent engendrer des différences d'expression génique ou de dynamique de la régulation du génome (comme la réparation de l'ADN par exemple). Notre approche est de lire des épigénomes naturels dans des organismes modèles (comme la levure) qui représentent des individus 'similaires mais non apparentés' et d'effectuer des expérimentations qui permettront d'associer certaines différences épigénomiques à certaines différences physiologiques.
La lecture des épigénomes et leur association à des caractères physiologiques requièrent le développement d'outils informatiques permettant l'exploitation des données expérimentales (telles que produites par ChIP-seq) et le développement d'approches statistiques qui permettront de transposer les concepts de la génétique quantitative à cette nouvelle discipline.
Pour plus de details, merci de consulter nos publications relatives à la découverte et aux caractéristiques des Single Nucleosome Epi-Polymorphisms.


 Français
Français  English (UK)
English (UK)