L’embryologie comparée ressuscitée par les nouvelles techniques de séquençage.
Des chercheuses du Laboratoire de biologie et de modélisation de la cellule de l’Ecole Normale Supérieure de Lyon, démontrent que la comparaison des transcriptomes permet de réconcilier l’analyse moléculaire des réseaux de gènes et l’observation histologique de l’embryologie classique. En fournissant des informations sur la régulation des gènes et l’abondance relative des types cellulaires dans les organes comparés, cette nouvelle approche permet de comprendre comment des profils de régulation génique très similaires peuvent conduire à des morphologies finales très différentes. Cette étude a été publiée le 15 février 2017 dans la revue Genome Biology.
La biologie du développement a longtemps été une science descriptive. Des générations d’embryologistes, l’œil rivé au microscope, ont décrit et comparé minutieusement la forme et la structure des embryons pour comprendre comment les organes sont mis en place ou comment les espèces diffèrent entre elles. Avant la fin du XXième siècle, ils ont été supplantés par une avant-garde de généticiens qui décortiquent le rôle de quelques gènes clés dans les processus de développement. De leurs expériences, ils tirent de précieux schémas sur la façon dont les gènes s’influencent les uns les autres. Mais les cellules, les tissus, le temps, et tous les autres gènes, soit tout ce qui fait le développement, en sont le plus souvent absents !
Depuis quelques années, les techniques qui permettent de quantifier simultanément le niveau des ARN transcrits (transcriptome) à partir de tous les gènes d’un génome se sont améliorées et démocratisées. Il devient possible de suivre et de corréler le développement morphologique d’un organe ou d’un embryon avec une série temporelle de transcriptomes. Du temps, et l’ensemble des gènes, cela peut apparaître comme un bonne approche, mais, en règle générale, seules les données d’expression de quelques gènes clés sont extraits de ces études de transcriptome, le reste étant considéré comme du bruit ininterprétable.
Sophie Pantalacci et Marie Sémon ont voulu savoir si c’était vraiment le cas, ou si ces très gros jeux de données sont actuellement sous-exploités. Elles ont comparé la dynamique d’expression de l’ensemble des gènes pendant le développement des molaires inférieure et supérieure de souris. Leurs dynamiques, et donc leurs programmes de développement, apparaissent globalement similaires, comme attendu pour deux organes sériels. Au-delà de cette similarité, les chercheuses ont identifié des différences, ou “signatures transcriptomiques, portées par des cohortes entières de gènes surexprimés dans l’une ou l’autre des molaires en développement. Elles ont montré que ces différences d’expression traduisent des différences de proportions des différents types cellulaires qui composent le germe dentaire, la surabondance d’un type cellulaire faisant apparaitre les gènes exprimés comme surexprimés à l’échelle de l’organe. Parmi ces différences de proportions cellulaires, certaines sont observées très tôt dans le développement et d’autres sont au contraire transitoires et marquent un évènement particulier de la morphogenèse. Ensemble, elles permettent d’expliquer comment un profil de régulation génique très similaire peut conduire à des morphologies finales très différentes.
En conclusion, un organe, des tissus, des cellules, du temps, l’ensemble des gènes, tout est réuni pour une nouvelle forme d’embryologie comparée, applicable à la comparaison des espèces entre elles : le « microscope moléculaire ».
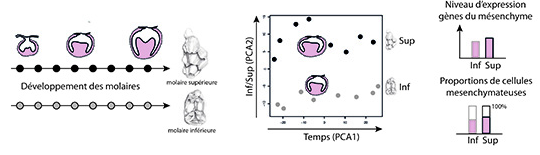
Figure : La comparaison des transcriptomes de différents organes, ou stades de développement, peut révéler les différences d’abondance des types cellulaires qui constituent ces organes. A gauche: comparaison des séries de transcriptomes pris au cours du développement de la molaire supérieure (sup, noir) et inférieure (inf, gris) de souris illustré à trois stades caractéristiques. Au centre : l’analyse multivariée des deux séries transcriptomiques révèle un axe de variation commun aux deux molaires, ordonnant les échantillons en fonction de leur âge, ce qui traduit une forte similarité de la régulation temporelle de l’expression. Le second axe qui oppose les deux molaires est dû à un excès relatif de tissu mésenchymateux dans la molaire supérieure par rapport à la molaire inférieure. A droite : les gènes exprimés dans le mésenchyme apparaissent en général surexprimés dans le germe supérieur. Cette différence d’expression faible, mais portée par un très grand nombre de gènes, correspond en réalité au fait que le germe supérieur est constitué d’une plus grande proportion de mésenchyme. La quantité de mésenchyme étant expérimentalement corrélée au nombre de cuspides formées, cette observation pourrait expliquer que la molaire supérieure forme davantage de cuspides (8 contre 6).
© Sophie Pantalacci - Marie Sémon
Transcriptomic signatures shaped by cell proportions shed light on comparative developmental biology.
Pantalacci S, Guéguen L, Petit C, Lambert A, Peterkovà R, Sémon M.
Genome Biol. 2017 Feb 15;18(1):29. doi: 10.1186/s13059-017-1157
