Bioinformatics
Welcoming bioinformatics students
We encourage bioinformatics students to contact us for a possible internship within our team. Our research subjects are at the interface between genome organization and RNA metabolism, in particular alternative splicing in humans. If you decide to join us you will be able to acquire knowledge in these biological fields.
In a UNIX/LINUX environment and according to the subject and the duration of your internship, you may have to process high-throughput sequencing data of public origin (i.e. ENCODE or GEO omnibus) or generated by our team. It can come from various experiments such as RNA-seq, ChIP-seq, Ribo-seq or 4C. So, You will develop skills on the tools to use for their processing (trimming, mapping, quality control, etc.). Because of the large size of these data sets (big data), you will be able to familiarise yourself with the use of grids.
Once processed, these data will need to be analysed. According to their nature, you will use existing bioinformatics tools or develop new ones using a programming language of your choice. In order to facilitate their exploration, these data must be stored and organised in the form, for example, of a database. In addition, a web-based interface may be developed.
Our team is composed of both biologists and bioinformaticians. So, you will be within a team where the exchange is important. Our team is attached to the LBMC (“Laboratoire de Biologie et Modélisation de la Cellule”) bringing together transdisciplinary research themes (Mathematics, Physics, etc.). Each team communicates on their work every Friday through seminars.
The LBMC has set up a biocomputing hub with the aim to set good bioinformatics practices and to train laboratory members. If you join us, you can be trained to use a version manager, containerization, virtualization and pipeline management tools. At the end of your internship and these training courses, you will have a solid base of skills.
Tools developed by the team :
- FaRLine :
This tool allows to analyse alternative splicing from RNAseq. (http://kissplice.prabi.fr/pipeline_ks_farline/)
Complementarity of Assembly-First and Mapping-First Approaches for Alternative Splicing Annotation and Differential Analysis From RNAseq Data (Mar 2018, Sci Rep) Clara Benoit-Pilven, et al. doi: 10.1038/s41598-018-217707-7
- Exon Ontology :
This interface allows to predict the functional consequences of variations in alternative splicing. Users can analyse exons to identify protein features that are associated with those exons.
Identification of Protein Features Encoded by Alternative Exons Using Exon Ontology ( Jun 2017, Gen. Research) Léon-Charles Tranchevent, et al. doi : 10.1101/gr.212696.116
- FasterDB :
It is a web-based interface linked to a database that describes known splice variants of human and murine protein-coding genes and their splicing pattern. The website also provides detailed information on transcripts and UTRs, including the strength of splice sites as well as miRNA binding sites. It is also possible to check the perservation between human and mouse genes at the exon level.
Endothelial, Epithelial, and Fibroblast Cells Exhibit Specific Splicing Programs Independently of Their Tissue of Origin ( Mar 2014, Gen. Research ) Pierre Mallinjoud, et al. doi : 10.1101/gr.162933.113
Recent publications :
Modulation of alternative splicing during early infection of human primary B lymphocytes with Epstein-Barr virus (EBV): a novel function for the viral EBNA-LP protein. Manet E, Polvèche H, Mure F, Mrozek-Gorska P, Roisné-Hamelin F, Hammerschmidt W, Auboeuf D, Gruffat H. NAR. Sept 2021. doi : 10.1093/nar/gkab787
SISTEMA: A large and standardized collection of transcriptome data sets for human pluripotent stem cell research. Jarrige M*, Polvèche H*, Carteron A, Janczarski S, Peschanski M, Auboeuf D, Martinat C. iScience. Jun 2021. doi : 10.1016/j.isci.2021.102767. http://sistema.ens-lyon.fr
Integrative Cell Type-Specific Multi-Omics Approaches Reveal Impaired Programs of Glial Cell Differentiation in Mouse Culture Models of DM1. González-Barriga A, Lallemant L, Dincã DM, Braz SO, Polvèche H, Magneron P, Pionneau C, Huguet-Lachon A, Claude JB, Chhuon C, Chiara Guerrera I, Bourgeois CF, Auboeuf D, Gourdon G and Gomes-Pereira M. Front. Cell. Neurosci. May 2021. doi: 10.3389/fncel.2021.662035
Intragenic Recruitment of NF-κB Drives Splicing Modifications Upon Activation by the Oncogene Tax of HTLV-1. Ben Ameur L, Marie P, Thenoz M, Giraud G, Combe E, Claude JB, Lemaire S, Fontrodona N, Polveche H, Bastien M, Gessain A, Wattel E, Bourgeois CF, Auboeuf D, Mortreux F. Nature Comm. jun 2021. doi: 10.1038/s41467-020-16853-x
Characterizing the interplay between gene nucleotide composition bias and splicing. Lemaire S, Fontrodona N, Aubé F, Claude JB, Polvèche H, Modolo L, Bourgeois CF, Mortreux F, Auboeuf D. Genome Biol. 2019 Nov 29;20(1):259. doi: 10.1186/s13059-019-1869-y. PMID: 31783898 Free PMC Article
Interplay between coding and exonic splicing regulatory sequences. Fontrodona N, Aubé F, Claude JB, Polvèche H, Lemaire S, Tranchevent LC, Modolo L, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2019 May;29(5):711-722. doi: 10.1101/gr.241315.118. Epub 2019 Apr 8. PMID: 30962178
Complementarity of assembly-first and mapping-first approaches for alternative splicing annotation and differential analysis from RNAseq data. Benoit-Pilven C, Marchet C, Chautard E, Lima L, Lambert MP, Sacomoto G, Rey A, Cologne A, Terrone S, Dulaurier L, Claude JB, Bourgeois CF, Auboeuf D, Lacroix V. Sci Rep. 2018 Mar 9;8(1):4307. doi: 10.1038/s41598-018-21770-7. PMID: 29523794 Free PMC Article
Identification of protein features encoded by alternative exons using Exon Ontology. Tranchevent LC, Aubé F, Dulaurier L, Benoit-Pilven C, Rey A, Poret A, Chautard E, Mortada H, Desmet FO, Chakrama FZ, Moreno-Garcia MA, Goillot E, Janczarski S, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2017 Jun;27(6):1087-1097. doi: 10.1101/gr.212696.116. Epub 2017 Apr 18. PMID: 28420690 Free
Endothelial, epithelial, and fibroblast cells exhibit specific splicing programs independently of their tissue of origin. Mallinjoud P, Villemin JP, Mortada H, Polay Espinoza M, Desmet FO, Samaan S, Chautard E, Tranchevent LC, Auboeuf D. Genome Res. 2014 Mar;24(3):511-21. doi: 10.1101/gr.162933.113. Epub 2013 Dec 4.
Former bioinformaticians of the team at the LBMC :
- Jean-Baptiste Claude (IR)
- Sébastien Lemaire (Doctorant)
- Léon-Charles Tranchevent (Post-doctorant)
- Arnaud Poret (Post-doctorant)
- Clara Benoit-Pilven (Doctorant)
- Émilie Chautard (Post-doctorant)
 Nicolas Fontrodona
Nicolas Fontrodona
Ingénieur d'études en Bio-informatique. ENS de Lyon – LBMC – CNRS.
Parcours et projets :
Après avoir effectué une licence en Modélisation et Informatique du Vivant (MIV) à Lyon, j’ai poursuivi mes études à Rennes ou j’ai obtenu le master de Biologie, Informatique et Génomique. Depuis novembre 2016, je suis ingénieur d’étude en bioinformatique dans l’équipe ReGArDS.
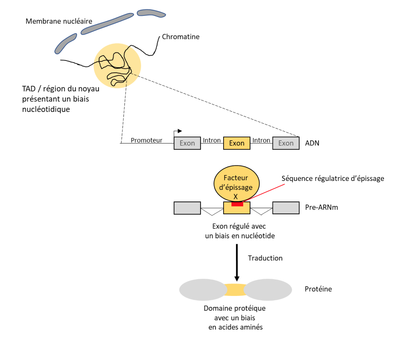
Mes missions principales sont centrées sur l’étude de la relation entre le biais de composition en nucléotide des exons et la régulation de leurs reconnaissances au cours de l’épissage alternatif. Plus précisément, avec mon équipe nous avons observé que les exons régulés par un même facteur d’épissage partageaient des biais de composition en nucléotide similaires. De plus, ces exons sont traduits en peptides qui eux-mêmes sont composés d’acides aminés aux propriétés physico-chimiques similaires. Ainsi, la régulation de l’inclusion d’exons par un facteur d’épissage va plus ou moins affecter la production de régions protéiques ayant des propriétés et donc potentiellement des fonctions similaires. Ces résultats ont été publiés dans Genome Research (Fontrodona et al. 2019). Ensuite, nous avons étendu l’analyse du biais de composition en nucléotide dans les exons régulés par un facteur d’épissage à leur région environnante tout en augmentant le nombre de facteurs d’épissages étudiés. Ceci a permis d’identifier deux groupes de facteurs : ceux augmentant l’inclusion d’exons riches en GC et ceux augmentant l’inclusion des exons riches en AT. Nous avons ensuite caractérisé deux mécanismes de régulation distincts s’exerçant pour l’épissage des exons régulés par ces deux groupes. Ces travaux ont été publiés dans Genome Biology (Lemaire et al. 2019). Actuellement, nous travaillons sur des jeux de données publiques issus de ChIA-PET pour construire une base données. Cette dernière nous permettra de tester si des exons partageant le même biais en nucléotide ou régulés par un même facteur d’épissage se trouvent à proximité spatiale dans le noyau.
Profil :
* Compétences :
- Langages de programmation : Python, Bash, R, SQL, HTML, CSS, Javascript
- OS : Linux, Windows, HPCC Grid Engine (SGE)
- Technologies de conteneurisation : Docker & Singularity
- Framework :
- workflow : Nextflow
- web : Django, VueJs, NuxtJS
* Missions :
– Analyse de données RNA-seq, ChIA-PET et Ribo-Seq
– Développement de pipeline d’analyses bio-informatiques
– Création et gestion de base de données
– Analyses statistiques
Publications :
- Interplay between coding and exonic splicing regulatory sequences. Fontrodona N, Aubé F, Claude JB, Polvèche H, Lemaire S, Tranchevent LC, Modolo L, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2019 May;29(5):711-722. doi: 10.1101/gr.241315.118. Epub 2019 Apr 8. PMID: 30962178
- Characterizing the interplay between gene nucleotide composition bias and splicing. Lemaire S, Fontrodona N, Aubé F, Claude JB, Polvèche H, Modolo L, Bourgeois CF, Mortreux F, Auboeuf D. Genome Biol. 2019 Nov 29;20(1):259. doi: 10.1186/s13059-019-1869-y. PMID: 31783898 Free PMC Article
Financements :
Institut National du Cancer (INCa)
Agence Nationale de la Recherche (ANR)
 Audrey Lapendry
Audrey Lapendry
PhD student in Bio-informatics. ENS de Lyon – LBMC.
Experience and projects :
Since October 2020, thanks to a “AFM-Téléthon” funding, I am a PhD student in Bioinformatics in the ReGArDS team, under the supervision of Didier Auboeuf. I did my further studies at the “Université Clermont Auvergne”, where I obtained a Bachelor's degree in Cellular Biology and Physiology and a Master's degree in Bioinformatics, Data Analysis and Modelling course.
Many neuromuscular diseases are associated with defects in both gene expression regulation and the cellular metabolism. However, the relationship between these two cellular processes is poorly known. The objective of my thesis is to test whether there is a direct link between amino acid metabolism, more specifically their bioavailability, and the regulation of gene expression, in particular alternative splicing that plays a major role in the differentiation of muscle cells. The results obtained could open new perspectives in terms of understanding the evolution of neuromuscular diseases, but also in terms of therapies that could for example be associated with the intake of particular amino acids through diet.
Before starting my PhD, I spent a year as an engineer within the ReGArDS team. My research work consisted of using and analysing ChIA-PET data. This technology allows to study chromatin interactions at the genome scale. We used this type of data to obtain the lists of exons and genes in interaction, which we stored in a database, in order to facilitate their exploration. Our next objective was to understand why these regions interact. For this, we considered, thanks to statistical analysis, several hypotheses: shared composition biases, regulation by the same splicing factor, etc. This project is still currently under study.
Profile:
* Skills:
* Programming languages: Python, Bash, R, SQL, HTML, PHP and JavaScript
* OS / Job schedulers: Linux, Windows & Mac / SGE and Slurm
* Workflow : Nextflow
* Code version manager: Git
* Experience:
– Data analysis: ChIP-seq, Hi-C and ChIA-PET
– Use and production of tools for the detection of no-B DNA structures
– Development of bioinformatics analysis pipelines
– Implementation of databases – Development of web interfaces – Statistical analysis
Funding: Association Française contre les Myopathies-Téléthon (AFM-Téléthon) 
 Hélène Polvèche
Hélène Polvèche
Ingénieur d'études en Bio-informatique. IStem – CECS
Laboratoire d'accueil : ReGarDS - LBMC - ENS de Lyon.
Créée en 2014 au sein de l’Institut des cellules Souches pour le Traitement et l’Étude des Maladies monogéniques (I-Stem) à Evry, la plateforme NGS & analyses génomiques (https://www.istem.eu/plateformes-technologiques/analysesgenomiques/) est une structure collaborative ouverte aux équipes académiques et a été récemment labellisée Genopole. La plateforme a permis depuis sa création de séquencer différents transcriptomes et d’aider des équipes dans l’analyse de ces projets (Darville et al, EBioMedicine 2016; Cambon et al, Molecular therapy. Methods & clinical development 2017; Galvan et al, Brain 2018; Hoch et al, Sci Rep. 2019).
Depuis 2015, la plateforme est en collaboration avec l’équipe ReGarDS afin de pérenniser la veille bio-informatique en constante évolution et d’explorer de façon poussée des données issues de séquençage. Ce travail collaboratif a notamment permis l’étude avec l’équipe de C. Martinat, de l’impact de l’épissage alternatif dans la dystrophie myotonique de type 1 (DM1) et d’identifier de nouvelles cibles thérapeutiques (Laustriat et al, Mol Ther Nucleic Acids. 2016, Maury et al, iScience. 2019).
En parallèle de ces projets, la plateforme développe de nouveaux outils de séquençage ou d’analyses bio-informatiques. Par exemple, sont en développement une base de données d’expression génique permettant d’étudier l’expression d’un ou de plusieurs gènes dans différents modèles cellulaires dérivés de cellules souches pluripotentes humaines (https://www.istem.eu/wp-content/uploads/2019/07/Poster_JOBIM2019_2019-06-25_hpolveche.pdf) ou encore une base de données recensant l’effet de l’épissage alternatif de plus d’une 50aine de facteurs d’épissage.
Profil :
* Missions :
- Traitements & Analyses de données (Transcriptomique) issues de NGS RNA-seq / Ampli-seq / Quant-seq (3'-seq) / miRNA-seq
Données de séquençage : Illumina / Ion Proton
- Mise en place de Pipelines d'analyses bio-informatiques
- Création d'outils via interfaces web
* Langages de programmation :
Bash, Perl, Python, R, HTML, SQL, PHP, Javascript, jQuery
* Supports / OS :
HPCC Grid Engine (SGE), Linux Debian - Ubuntu – Windows
Publications :
- Characterizing the interplay between gene nucleotide composition bias and splicing. (Nov. 2019, Gen. Biology) Lemaire S, et al. doi: 10.1186/s13059-019-1869-y
- Physical and functional interaction between SET1/COMPASS complex component CFP-1 and a Sin3S HDAC complex in C. elegans. (Oct. 2019, NAR) Beurton F, et al. doi: 10.1093/nar/gkz880
- Interplay between coding and exonic splicing regulatory sequences. (May 2019, Gen. Research) Fontrodona N, Aubé F, et al. doi: 10.1101/gr.241315.118
- Human pluripotent stem cell-based drug screening reveals therapeutic potential of repurposable cardiac glycosides in myotonic dystrophy type 1 (Dec. 2018, iScience) Yves Maury, et al. doi :10.1016/j.isci.2018.12.019
