Genome organization, processing and fate of RNA - Didier Auboeuf

Chef de projet : Didier Auboeuf
L’expression des gènes est classiquement représentée comme une succession d’étapes successives et indépendantes (la transcription, l’épissage, l’export et la traduction). Cependant, chacune de ces étapes est couplée à l’étape d’avant et à celle d’après. Ce couplage physique entre étapes implique qu’une étape de l’expression des gènes peut avoir de multiples conséquences sur les autres. C’est dans ce contexte que l’équipe s’intéresse au rôle de ce couplage dans la coordination de la régulation de l’expression des gènes et donc dans l’adaptation physiologique des cellules à des variations de leur environnement.
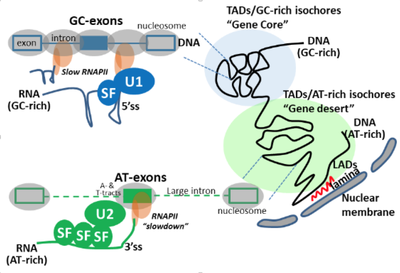
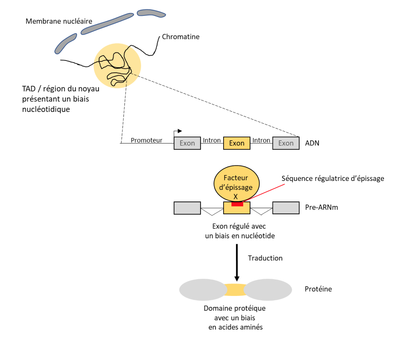
Le groupe de Didier Auboeuf s’intéresse notamment au biais de composition nucléotidiques des gènes et à la conséquence de ces biais de composition à la fois sur l’organisation du génome, l’activité transcriptionnelle des gènes et la maturation (épissage) des transcrits. Le groupe s’intéresse également à l’impact de biais de composition en nucléotides des gènes sur les biais de composition en acides aminés des protéines issues de ces gènes ; ces biais de composition en acides aminés ayant des conséquences sur les propriétés physicochimiques des protéines et donc leurs activités. Une hypothèse est que les biais de composition nucléotidiques de différentes régions génomiques génèrent un continuum de l’organisation en 3D du génome à l’interactome des protéines.
Une deuxième activité du groupe concerne la compréhension des mécanismes d’adaptation des cellules cancéreuses à des stress et notamment aux thérapies anti-cancéreuses. En effet, toutes les thérapies anti-cancéreuses sont associées à l’émergence de clones cellulaires résistants. Pour adresser cette problématique, le groupe utilise différents modèles cellulaires cancéreux et adresse le lien entre métabolisme, en particulier le métabolisme des acides aminés et l’adaptabilité des cellules cancéreuses à des stress. Une hypothèse est qu’il devrait être possible d’empêcher l’émergence de clones résistants en bloquant l’adaptabilité des cellules cancéreuses via leur métabolisme.
Les approches du groupe sont basées sur l’utilisation et l’exploitation de données à large échelles (transcriptomique, HiC, ChIP…). Ces données sont publiques ou générées par l’équipe. A partir de l’analyse de ces données expérimentales à large échelle, le groupe génère des modèles qui sont challengés expérimentalement.
 Nicolas Fontrodona
Nicolas Fontrodona
Ingénieur d'études en Bio-informatique. ENS de Lyon – LBMC – CNRS.
Parcours et projets :
Après avoir effectué une licence en Modélisation et Informatique du Vivant (MIV) à Lyon, j’ai poursuivi mes études à Rennes ou j’ai obtenu le master de Biologie, Informatique et Génomique. Depuis novembre 2016, je suis ingénieur d’étude en bioinformatique dans l’équipe ReGArDS.
Mes missions principales sont centrées sur l’étude de la relation entre le biais de composition en nucléotide des exons et la régulation de leurs reconnaissances au cours de l’épissage alternatif. Plus précisément, avec mon équipe nous avons observé que les exons régulés par un même facteur d’épissage partageaient des biais de composition en nucléotide similaires. De plus, ces exons sont traduits en peptides qui eux-mêmes sont composés d’acides aminés aux propriétés physico-chimiques similaires. Ainsi, la régulation de l’inclusion d’exons par un facteur d’épissage va plus ou moins affecter la production de régions protéiques ayant des propriétés et donc potentiellement des fonctions similaires. Ces résultats ont été publiés dans Genome Research (Fontrodona et al. 2019). Ensuite, nous avons étendu l’analyse du biais de composition en nucléotide dans les exons régulés par un facteur d’épissage à leur région environnante tout en augmentant le nombre de facteurs d’épissages étudiés. Ceci a permis d’identifier deux groupes de facteurs : ceux augmentant l’inclusion d’exons riches en GC et ceux augmentant l’inclusion des exons riches en AT. Nous avons ensuite caractérisé deux mécanismes de régulation distincts s’exerçant pour l’épissage des exons régulés par ces deux groupes. Ces travaux ont été publiés dans Genome Biology (Lemaire et al. 2019). Actuellement, nous travaillons sur des jeux de données publiques issus de ChIA-PET pour construire une base données. Cette dernière nous permettra de tester si des exons partageant le même biais en nucléotide ou régulés par un même facteur d’épissage se trouvent à proximité spatiale dans le noyau.
Profil :
* Compétences :
- Langages de programmation : Python, Bash, R, SQL, HTML, CSS, Javascript
- OS : Linux, Windows, HPCC Grid Engine (SGE)
- Technologies de conteneurisation : Docker & Singularity
- Framework :
- workflow : Nextflow
- web : Django, VueJs, NuxtJS
* Missions :
– Analyse de données RNA-seq, ChIA-PET et Ribo-Seq
– Développement de pipeline d’analyses bio-informatiques
– Création et gestion de base de données
– Analyses statistiques
Publications :
- Interplay between coding and exonic splicing regulatory sequences. Fontrodona N, Aubé F, Claude JB, Polvèche H, Lemaire S, Tranchevent LC, Modolo L, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2019 May;29(5):711-722. doi: 10.1101/gr.241315.118. Epub 2019 Apr 8. PMID: 30962178
- Characterizing the interplay between gene nucleotide composition bias and splicing. Lemaire S, Fontrodona N, Aubé F, Claude JB, Polvèche H, Modolo L, Bourgeois CF, Mortreux F, Auboeuf D. Genome Biol. 2019 Nov 29;20(1):259. doi: 10.1186/s13059-019-1869-y. PMID: 31783898 Free PMC Article
Financements :
Institut National du Cancer (INCa)
Agence Nationale de la Recherche (ANR)
 Audrey Lapendry
Audrey Lapendry
PhD student in Bio-informatics. ENS de Lyon – LBMC.
Experience and projects :
Since October 2020, thanks to a “AFM-Téléthon” funding, I am a PhD student in Bioinformatics in the ReGArDS team, under the supervision of Didier Auboeuf. I did my further studies at the “Université Clermont Auvergne”, where I obtained a Bachelor's degree in Cellular Biology and Physiology and a Master's degree in Bioinformatics, Data Analysis and Modelling course.
Many neuromuscular diseases are associated with defects in both gene expression regulation and the cellular metabolism. However, the relationship between these two cellular processes is poorly known. The objective of my thesis is to test whether there is a direct link between amino acid metabolism, more specifically their bioavailability, and the regulation of gene expression, in particular alternative splicing that plays a major role in the differentiation of muscle cells. The results obtained could open new perspectives in terms of understanding the evolution of neuromuscular diseases, but also in terms of therapies that could for example be associated with the intake of particular amino acids through diet.
Before starting my PhD, I spent a year as an engineer within the ReGArDS team. My research work consisted of using and analysing ChIA-PET data. This technology allows to study chromatin interactions at the genome scale. We used this type of data to obtain the lists of exons and genes in interaction, which we stored in a database, in order to facilitate their exploration. Our next objective was to understand why these regions interact. For this, we considered, thanks to statistical analysis, several hypotheses: shared composition biases, regulation by the same splicing factor, etc. This project is still currently under study.
* Skills:
* Programming languages: Python, Bash, R, SQL, HTML, PHP and JavaScript
* OS / Job schedulers: Linux, Windows & Mac / SGE and Slurm
* Workflow : Nextflow
* Code version manager: Git
* Experience:
– Data analysis: ChIP-seq, Hi-C and ChIA-PET
– Use and production of tools for the detection of no-B DNA structures
– Development of bioinformatics analysis pipelines
– Implementation of databases – Development of web interfaces – Statistical analysis
Funding: Association Française contre les Myopathies-Téléthon (AFM-Téléthon)
Formers bio-informaticians of the team in lbmc :
- Jean-Baptiste Claude (IR)
- Sébastien Lemaire (Doctorant)
- Léon-Charles Tranchevent (Post-doctorant)
- Arnaud Poret (Post-doctorant)
- Clara Benoit-Pilven (Doctorant)
- Émilie Chautard (Post-doctorant)
in silico tools of the team :
- FaRLine :
(http://fasterdb.ens-lyon.fr/FARLINE_human_mouse_2018.tgz)
Cet outil permet d’analyser l’épissage alternatif à partir de RNAseq. (http://kissplice.prabi.fr/pipeline_ks_farline/)
Complementarity of Assembly-First and Mapping-First Approaches for Alternative Splicing Annotation and Differential Analysis From RNAseq Data (Mar 2018, Sci Rep) Clara Benoit-Pilven, et al. doi: 10.1038/s41598-018-217707-7
- Exon Ontology :
(http://fasterdb.ens-lyon.fr/ExonOntology/index.php)
Cette interface permet de prévoir les conséquences fonctionnelles des variations d'épissages alternatifs. Les utilisateurs peuvent analyser les exons pour identifier les caractéristiques des protéines qui sont associées à ces exons.
Identification of Protein Features Encoded by Alternative Exons Using Exon Ontology ( Jun 2017, Gen. Research) Léon-Charles Tranchevent, et al. doi : 10.1101/gr.212696.116
- FasterDB :
(http://fasterdb.ens-lyon.fr/faster/home.pl)
Il s’agit d’une interface web vers une base de données qui décrit les variants d'épissage connus des gènes codant pour les protéines humaines et murines et leur schéma d'épissage. Le site web fournit également des informations détaillées sur les transcrits et les UTR, y compris la force des sites d'épissage ainsi que les sites de liaison des miARN. Il est également possible de vérifier la conservation entre les gènes humains et ceux de souris au niveau des exons.
Endothelial, Epithelial, and Fibroblast Cells Exhibit Specific Splicing Programs Independently of Their Tissue of Origin ( Mar 2014, Gen. Research ) Pierre Mallinjoud, et al. doi : 10.1101/gr.162933.113
 Hélène Polvèche
Hélène Polvèche
Ingénieur d'études en Bio-informatique. IStem – CECS
Créée en 2014 au sein de l’Institut des cellules Souches pour le Traitement et l’Étude des Maladies monogéniques (I-Stem) à Evry, la plateforme NGS & analyses génomiques (https://www.istem.eu/plateformes-technologiques/analysesgenomiques/) est une structure collaborative ouverte aux équipes académiques et a été récemment labellisée Genopole. La plateforme a permis depuis sa création de séquencer différents transcriptomes et d’aider des équipes dans l’analyse de ces projets (Darville et al, EBioMedicine 2016; Cambon et al, Molecular therapy. Methods & clinical development 2017; Galvan et al, Brain 2018; Hoch et al, Sci Rep. 2019).
Depuis 2015, la plateforme est en collaboration avec l’équipe ReGarDS afin de pérenniser la veille bio-informatique en constante évolution et d’explorer de façon poussée des données issues de séquençage. Ce travail collaboratif a notamment permis l’étude avec l’équipe de C. Martinat, de l’impact de l’épissage alternatif dans la dystrophie myotonique de type 1 (DM1) et d’identifier de nouvelles cibles thérapeutiques (Laustriat et al, Mol Ther Nucleic Acids. 2016, Maury et al, iScience. 2019).
En parallèle de ces projets, la plateforme développe de nouveaux outils de séquençage ou d’analyses bio-informatiques. Par exemple, sont en développement une base de données d’expression génique permettant d’étudier l’expression d’un ou de plusieurs gènes dans différents modèles cellulaires dérivés de cellules souches pluripotentes humaines (https://www.istem.eu/wp-content/uploads/2019/07/Poster_JOBIM2019_2019-06-25_hpolveche.pdf) ou encore une base de données recensant l’effet de l’épissage alternatif de plus d’une 50aine de facteurs d’épissage.
Profil :
* Missions :
- Traitements & Analyses de données (Transcriptomique) issues de NGS RNA-seq / Ampli-seq / Quant-seq (3'-seq) / miRNA-seq
Données de séquençage : Illumina / Ion Proton
- Mise en place de Pipelines d'analyses bio-informatiques
- Création d'outils via interfaces web
* Langages de programmation :
Bash, Perl, Python, R, HTML, SQL, PHP, Javascript, jQuery
* Supports / OS :
HPCC Grid Engine (SGE), Linux Debian - Ubuntu – Windows
Publications :
- Characterizing the interplay between gene nucleotide composition bias and splicing. (Nov. 2019, Gen. Biology) Lemaire S, et al. doi: 10.1186/s13059-019-1869-y
- Physical and functional interaction between SET1/COMPASS complex component CFP-1 and a Sin3S HDAC complex in C. elegans. (Oct. 2019, NAR) Beurton F, et al. doi: 10.1093/nar/gkz880
- Interplay between coding and exonic splicing regulatory sequences. (May 2019, Gen. Research) Fontrodona N, Aubé F, et al. doi: 10.1101/gr.241315.118
- Human pluripotent stem cell-based drug screening reveals therapeutic potential of repurposable cardiac glycosides in myotonic dystrophy type 1 (Dec. 2018, iScience) Yves Maury, et al. doi :10.1016/j.isci.2018.12.019
 Fabien Aubé
Fabien Aubé
Doctorant en Biologie - ENS de Lyon - LBMC
Parcours et projet de thèse :
Après une classe prépa BCPST, j’ai intégré l’école d’Ingénieurs VetAgro-Sup de Clermont-Ferrand pour me spécialiser en génétique des plantes. Après l’obtention de mon diplôme, j’ai décroché un premier CDD d’un an en tant qu’ingénieur d’études à l’INRA de Versailles. J’ai ensuite décidé de changer de domaine et ai rejoint l’équipe de Didier Auboeuf en 2015 ou j’ai évolué en tant qu’ingénieur d’études pendant près de 5 ans, avant de débuter ma thèse fin 2019.
Mon projet de thèse est financé par la Région Auvergne Rhône Alpes
Une des découvertes majeures de ces dernières années concernant les cellules cancéreuses a été la mise en évidence de leur « capacité » à modifier leur phénotype de façon bien plus importante qu’initialement anticipé. La mise en évidence de la plasticité phénotypique des cellules cancéreuses permet de mieux comprendre pourquoi toutes les thérapies anti-tumorales développées jusqu’à présent, conduisent très fréquemment à l’émergence de clones résistants, c’est-à-dire de cellules qui ne répondent plus aux agents anti-tumoraux (phénomène d’échappement thérapeutique). Maîtriser, ou du moins contrecarrer la plasticité phénotypique des cellules cancéreuses, est l’enjeu majeur des thérapies anti-cancéreuses de nouvelles générations à développer.
Une deuxième découverte majeure de ces dernières années est la mise en évidence de modifications profondes du métabolisme des cellules cancéreuses. Ces modifications du métabolisme cellulaire sont au cœur du processus d’adaptation des cellules cancéreuses. En effet, les changements de l’activité métabolique des cellules cancéreuses impactent l’expression des gènes, et donc le phénotype cancéreux. Dans ce contexte, d’importants efforts ont été faits pour développer des thérapies ciblant le métabolisme cellulaire. Néanmoins, ces thérapies risquent, à l’instar de toutes les thérapies anti-cancéreuses, d’être inefficaces en raison de l’émergence de clones résistants. Afin de contrecarrer l’émergence de clones résistants, la stratégie qui semble à l’heure actuelle la plus appropriée consiste à combiner des thérapies.
L’objectif de mon projet fondé sur des approches à large échelle, des analyses bio-informatiques, de la modélisation et un crible semi-haut débit, est de développer une stratégie rationnelle visant à combiner des approches thérapeutiques dans le but ultime d’inhiber la prolifération des cellules cancéreuses tout en les empêchant de s’adapter aux thérapies. L’hypothèse principale de ce projet est que la déprivation d’au moins un acide aminé limitera la capacité des cellules cancéreuses à s’adapter à des thérapies anti-cancéreuses classiques.
Profil :
* Compétences :
-Biologie cellulaire
-Biologie moléculaire
-Programmation
Publications :
Translation-dependent and independent mRNA decay occur through mutually exclusive pathways that are defined by ribosome density during T Cell activation Blandine C. Mercier, Emmanuel Labaronne, David Cluet, Alicia Bicknell, Antoine Corbin, Laura Guiguettaz, Fabien Aubé, Laurent Modolo, Didier Auboeuf, Melissa J. Moore, Emiliano P. Ricci Under review
The Consequences of a Disruption in Cyto-Nuclear Coadaptation on the Molecular Response to a Nitrate Starvation in Arabidopsis. Chardon F, Cueff G, Delannoy E, Aubé F, Lornac A, Bedu M, Gilard F, Pateyron S, Rogniaux H, Gargaros A, Mireau H, Rajjou L, Martin-Magniette ML, Budar F. Plants (Basel). 2020 May 1;9(5):573. doi: 10.3390/plants9050573. PMID: 32369924
Characterizing the interplay between gene nucleotide composition bias and splicing. Lemaire S, Fontrodona N, Aubé F, Claude JB, Polvèche H, Modolo L, Bourgeois CF, Mortreux F, Auboeuf D. Genome Biol. 2019 Nov 29;20(1):259. doi: 10.1186/s13059-019-1869-y. PMID: 31783898
Interplay between coding and exonic splicing regulatory sequences. Fontrodona N, Aubé F, Claude JB, Polvèche H, Lemaire S, Tranchevent LC, Modolo L, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2019 May;29(5):711-722. doi: 10.1101/gr.241315.118. Epub 2019 Apr 8. PMID: 30962178
Genome editing in primary cells and in vivo using viral-derived Nanoblades loaded with Cas9-sgRNA ribonucleoproteins. Mangeot PE, Risson V, Fusil F, Marnef A, Laurent E, Blin J, Mournetas V, Massouridès E, Sohier TJM, Corbin A, Aubé F, Teixeira M, Pinset C, Schaeffer L, Legube G, Cosset FL, Verhoeyen E, Ohlmann T, Ricci EP. Nat Commun. 2019 Jan 3;10(1):45. doi: 10.1038/s41467-018-07845-z. PMID: 30604748
Three new pentatricopeptide repeat proteins facilitate the splicing of mitochondrial transcripts and complex I biogenesis in Arabidopsis. Wang C, Aubé F, Quadrado M, Dargel-Graffin C, Mireau H. J Exp Bot. 2018 Oct 12;69(21):5131-5140. doi: 10.1093/jxb/ery275. PMID: 30053059
Identification of protein features encoded by alternative exons using Exon Ontology. Tranchevent LC, Aubé F, Dulaurier L, Benoit-Pilven C, Rey A, Poret A, Chautard E, Mortada H, Desmet FO, Chakrama FZ, Moreno-Garcia MA, Goillot E, Janczarski S, Mortreux F, Bourgeois CF, Auboeuf D. Genome Res. 2017 Jun;27(6):1087-1097. doi: 10.1101/gr.212696.116. Epub 2017 Apr 18. PMID: 28420690
The pentatricopeptide repeat protein MTSF2 stabilizes a nad1 precursor transcript and defines the 3΄ end of its 5΄-half intron. Wang C, Aubé F, Planchard N, Quadrado M, Dargel-Graffin C, Nogué F, Mireau H. Nucleic Acids Res. 2017 Jun 2;45(10):6119-6134. doi: 10.1093/nar/gkx162. PMID: 28334831
Financements :

 Jélane SAAD-ABOUZAYD
Jélane SAAD-ABOUZAYD
Doctorante en Biologie du cancer. ENS de Lyon – LBMC.
Parcours et projet :
Depuis janvier 2019, je suis doctorante dans l’équipe ReGArDS, sous la supervision du Dr Didier Auboeuf. J’ai obtenu mon diplôme de Master à l’Université de Caen en PIDG (Physiologie, Immunité, Différenciation, Génétique), où j’ai choisi de me spécialiser en génétique du cancer. Mon intérêt pour la recherche dans le domaine du cancer m’a amené, tout au long de mon parcours universitaire, à effectuer des stages dans diverses structures telles que McGill Cancer center au Canada, ou encore l’Institut Gustave Roussy à Paris.
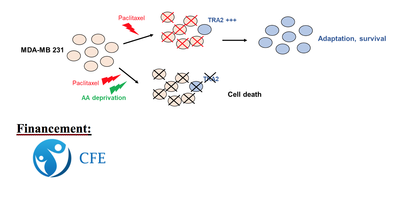
La résistance aux thérapies anti-cancéreuses est le principal enjeu de la lutte contre le cancer. Les thérapies ciblées augmentent la durée de vie des patients mais n’empêchent pas la survenue de récidives. Il est ainsi urgent de développer de nouvelles classes de médicaments qui prennent en compte « l’adaptabilité » et la plasticité des cellules cancéreuses aux traitements conventionnels comme la chimiothérapie ou aux thérapies ciblées.
Nous savons aujourd’hui qu’une des caractéristiques des cellules cancéreuses est la « reprogrammation » de leur métabolisme cellulaire. En particulier, le taux de prolifération élevé des cellules cancéreuses nécessite des modifications du métabolisme des acides aminés, pour satisfaire le besoin de traduction protéique. vC’est dans ce contexte que l’addiction des tumeurs à certains acides aminés spécifiques a été décrite. Par exemple, ne pouvant plus produire une quantité suffisante de glutamine, les cellules cancéreuses dépendent de leur capacité à les obtenir de leur propre environnement.
Sur la base de ces observations, il a été proposé qu'une diminution de la biodisponibilité de certains acides aminés peut être une stratégie thérapeutique anti-cancéreuse intéressante.
En outre, les facteurs d’épissage font partie des protéines essentielles à l’adaptation des cellules cancéreuses aux thérapies. L’objectif de ma thèse est de combiner la chimiothérapie avec une privation d’acide aminé pour empêcher la production du facteur d’épissage TRA2 (Transformer-2), et ainsi contourner la résistance des cellules de cancer mammaires (MDA-MB-231) au Paclitaxel.