Topologie de la chromatine, transcription, et épissage alternatif au cours des processus inflammatoires et oncogènes - Franck Mortreux
 Topologie de la chromatine, transcription, et épissage alternatif au cours des processus inflammatoires et oncogènes.
Topologie de la chromatine, transcription, et épissage alternatif au cours des processus inflammatoires et oncogènes.
Chef de projet : Franck Mortreux
Les mécanismes de régulation de l’épissage ont longtemps été considérés comme des évènements post-transcriptionnels dépendant exclusivement de l’abondance des facteurs d’épissage et de leur régulation post-traductionnelle. Néanmoins, il est aujourd’hui bien établi que ces processus sont finement couplés à la transcription pour la grande majorité des gènes humains. Les paramètres qui influencent les décisions d’épissage sont multiples incluant notamment le remodelage de la chromatine et la cinétique d’élongation de l’ARNPII mais également la structure des promoteurs, des enhancers, et la nature des facteurs (et/ou cofacteurs) de transcription qui y sont recrutés.
Nos travaux s’intéressent aux mécanismes moléculaires qui régulent la topologie de la chromatine, la transcription, et l'épissage alternatif au cours des processus inflammatoires et oncogènes. Notre modèle d'étude est l'infection par le virus oncogène HTLV-1 (Human T-cell leukemia Virus type 1) qui est responsable de nombreuses pathologies inflammatoires et d’une hémopathie maligne T-CD4+ très agressive (la Leucémie/Lymphome T de l’Adulte (ATLL)) qui se développent chez 2 à 5 % des individus infectés. Nous avons récemment découvert que, outre les nombreuses altérations transcriptionnelles qui accompagnent l’infection, des perturbations de l’épissage alternatif des transcrits cellulaires sont également fréquentes, plus particulièrement au stade ATLL. De façon intéressante, ces modifications affectent des voies fonctionnelles différentes de celles dérégulées au niveau transcriptionnel. Elles concernent notamment des processus d’adhésion cellulaire pouvant contribuer à l’extravasion des cellules infectées vers d’autres compartiments anatomiques, mais également d’autres voies fonctionnelles pouvant participer à la physiopathogénie de l’infection.
Les oncogènes viraux Tax et HBZ sont bien connus pour agir comme des co-facteurs transcriptionnels qui perturbent l’activité de nombreux régulateurs de la transcription cellulaire tels que NF-kB, AP-1, et SMAD. Ces modifications concernent essentiellement les gènes qui contrôlent la prolifération cellulaire, l’intégrité du génome et la réponse immune. Nos résultats récents indiquent que TAX et HBZ génèrent par aileurs de nombreuses modifications d’épissage en intervenant vraisemblablement sur les mécanismes de couplage transcription/épissage. Nous utilisons les propriétés de ces facteurs viraux pour disséquer les mécanismes qui relient la régulation de l’épissage alternatif au remodelage de la chromatine et à l’organisation spatio-temporelle des gènes.
Nos études combinent des approches expérimentales et bioinformatiques. Nous utilisons notamment les méthodes de RNA-seq, qChiP, ChiP-seq, Cut&Run, 3C (Chromosome Conformation Capture), 4C et Hi-C pour l’analyse des transcriptomes et de la chromatine. Nous avons par ailleurs développé les outils TALE, CRISPR/Cas9, CRISPR-dCas9-KRAB, CRISPR-dCas9-VPR, CLOuD9 pour des modifications expérimentales ciblées à l’ADN et à la chromatine.
Trois axes sont développés :
 Rôle de p65/RELA et de l’hélicase à ARN DDX17 dans la définition des cibles d’épissage lors de l’activation de la voie NF-kB par TAX. Lamya Ben Ameur (post-doc)
Rôle de p65/RELA et de l’hélicase à ARN DDX17 dans la définition des cibles d’épissage lors de l’activation de la voie NF-kB par TAX. Lamya Ben Ameur (post-doc)
Nous adressons ici le rôle de TAX dans les perturbations de régulation de l’épissage alternatif. Nos données montrent que ces nouvelles propriétés de l’oncogène viral s’opèrent indépendamment de ses effets sur l’expression des gènes. Plus particulièrement, nos résultats révèlent un mécanisme de régulation où le recrutement intragénique du facteur detranscription NF-kB RELA permet d’ancrer à la chromatine le facteur d’épissage DDX17 qui régule l’exon cible via son activité hélicase. De façon intéressante, ce mécanisme a été également observé lorsque la voie NF-kB est activée indépendamment de TAX. Ces données révèlent donc un mécanisme physiologique de définition de cibles d’épissage spécifique de la voie NF-kB, qui serait vraisemblablement exacerbé par TAX par ses effets constitutifs sur l’activation de la voie.
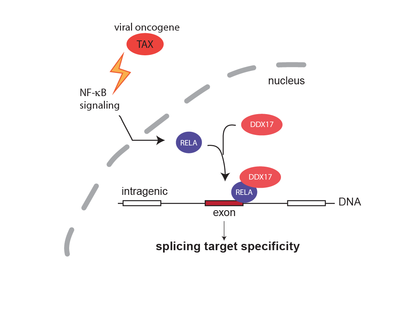
Sur la base de ces nouvelles données, nous nous intéressons dorénavant aux impacts fonctionnels de ces régulations d’épissage dans les processus d’inflammation et de transformation de la cellule, et nous explorons au plan moléculaire les mécanismes d’épissage alternatifs distinctement reliés aux 2 voies de signalisation de NF-kB, canoniques et non-canoniques.
 Coordination spatio-temporelle de l’épissage alternatif des gènes de réponse à la voie NF-kB : rôle de TAX . Paul Marie (PhD)
Coordination spatio-temporelle de l’épissage alternatif des gènes de réponse à la voie NF-kB : rôle de TAX . Paul Marie (PhD)
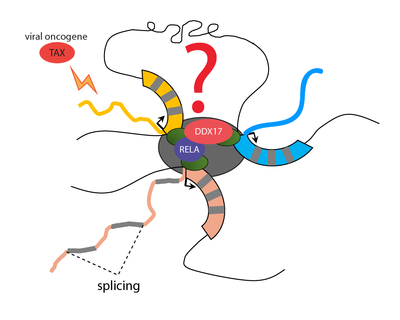 L’expression physiologique des gènes humains nécessite une coordination précise de la transcription des gènes. Il a été proposé que cette coordination spatiotemporelle s’opère dans des foci nucléaires identifiés comme des usines à transcription (transcription factories) composées de plusieurs RNAPII, de facteurs essentiels à l’initiation et à l’élongation transcriptionnelle, et des régulateurs post-transcriptionnels. Une des fonctions premières des « transcription factories » est le regroupement de gènes co-régulés par les facteurs de transcription d’une même famille. Par exemple, en réponse au TNF-α, des approches de 3C (Chromosome Conformation Capture) et de RNA-FISH ont permis d’identifier la formation de NF-kB factories où se coordonne la transcription de gènes répondeurs à l’activation de la voie NF-kB. Les facteurs NF-kB et les hélicases à ARN DDX5/17 ont été également directement impliqués dans la formation de boucle ADN au cours de processus transcriptionnels. L’oncogène viral TAX est également capable de s’organiser en structures nucléaires (TAX speckles) enrichies en facteurs NF-kB, en RNAPII hyperphosphorylées et en facteurs d’épissage. Dans ce contexte, nos objectifs sont d’adresser les effets de TAX sur la topologie de la chromatine, le regroupement de gènes et leur régulations transcriptionnelles et post-transcriptionnelles.
L’expression physiologique des gènes humains nécessite une coordination précise de la transcription des gènes. Il a été proposé que cette coordination spatiotemporelle s’opère dans des foci nucléaires identifiés comme des usines à transcription (transcription factories) composées de plusieurs RNAPII, de facteurs essentiels à l’initiation et à l’élongation transcriptionnelle, et des régulateurs post-transcriptionnels. Une des fonctions premières des « transcription factories » est le regroupement de gènes co-régulés par les facteurs de transcription d’une même famille. Par exemple, en réponse au TNF-α, des approches de 3C (Chromosome Conformation Capture) et de RNA-FISH ont permis d’identifier la formation de NF-kB factories où se coordonne la transcription de gènes répondeurs à l’activation de la voie NF-kB. Les facteurs NF-kB et les hélicases à ARN DDX5/17 ont été également directement impliqués dans la formation de boucle ADN au cours de processus transcriptionnels. L’oncogène viral TAX est également capable de s’organiser en structures nucléaires (TAX speckles) enrichies en facteurs NF-kB, en RNAPII hyperphosphorylées et en facteurs d’épissage. Dans ce contexte, nos objectifs sont d’adresser les effets de TAX sur la topologie de la chromatine, le regroupement de gènes et leur régulations transcriptionnelles et post-transcriptionnelles.
 Régulation de l’épissage rétrograde et de la biogenèse des ARN circulaires : rôle d'HBZ Julien Ladet (PhD)
Régulation de l’épissage rétrograde et de la biogenèse des ARN circulaires : rôle d'HBZ Julien Ladet (PhD)
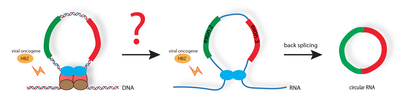
Les ARN circulaires (circRNAs) constituent une large classe d’ARN retrouvés fréquemment dérégulés dans les tumeurs. Leurs fonctions biologiques sont très diverses, incluant la séquestration de micro-ARN (miRNA sponges), la régulation transcriptionnelle, et la synthèse de peptides/protéines spécifiques. La régulation de l’expression des circRNA fait intervenir des processus d’épissage rétrogrades (back splicing) dont les mécanismes restent encore mal compris. L’analyse de l’abondance des circRNAs dans les ATL révèle de fortes perturbations des circRNA constitutifs (exprimés également dans les cellules CD4+ de donneurs), dont certains sont déjà définis comme des suppresseurs de tumeurs. Nos données préliminaires révèlent que l’oncogène viral HBZ, jusqu’à présent défini comme un régulateur transcriptionnel, interagit avec de nombreux régulateurs d’épissage et d’autres facteurs déjà décrits dans la biogenèse des circRNAs. Notre hypothèse est que certaines modifications de circRNA dans les ATL pourraient être associées aux effets d’HBZ sur des RBP qui régulent à la fois l’épissage alternatif et la topologie de l’ADN. Nos objectifs sont de disséquer les mécanismes moléculaires qui relient la conformation de la chromatine et la régulation de l’épissage alternatif à la biogenèse des circRNAs.
Publications récentes :
- Antony Ceraulo, Hélène Lapillonne, Meyling H Cheok, Claude Preudhomme, Hervé Dombret, Christine Terré, Juliette Lambert, Guy Leverger, Yves Bertrand, Franck Mortreux*, Eric Wattel*. Prognostic impact of ABCA3 expression in adult and pediatric acute myeloid leukemia: an ALFA-ELAM02 joint study. Blood Adv. 2022 Jan 10. doi: 10.1182/bloodadvances.2021006040.
- Ameur LB, Marie P, Thenoz M, Giraud G, Combe E, Claude JB, Lemaire S, Fontrodona N, Polveche H, Bastien M, Gessain A, Wattel E, Bourgeois CF, Auboeuf D, Mortreux F. Intragenic recruitment of NF-κB drives splicing modifications upon activation by the oncogene Tax of HTLV-1. Nat Commun. 2020 Jun 16;11(1):3045. doi: 10.1038/s41467-020-16853-x.
- Ladet J, Mortreux F. Circular RNA, actors and biomarkers of cancers. Med Sci (Paris). 2020 Oct;36(10):935-938. doi: 10.1051/medsci/2020165.
- Mohamed, A. M., Balsat, M., Koering, C., Maucort-Boulch, D., Boissel, N., Payen-Gay, L., Cheok, M., Mortada, H., Auboeuf, D., Pinatel, C., El-Hamri, M., Tigaud, I., Hayette, S., Dumontet, C., Cros, E., Flandrin-Gresta, P., Nibourel, O., Preudhomme, C., Thomas, X., Nicolini, F. E., Solly, F., Guyotat, D., Campos, L., Michallet, M., Ceraulo, A., *Mortreux, F., and *Wattel, E. (2017) TET2 exon 2 skipping is an independent favorable prognostic factor for cytogenetically normal acute myelogenous leukemia (AML): TET2 exon 2 skipping in AML. Leuk Res 56, 21-28
- Mohamed, A. M., Balsat, M., Thenoz, M., Koering, C., Payen-Gay, L., Cheok, M., Mortada, H., Auboeuf, D., Pinatel, C., El-Hamri, M., Dumontet, C., Cros, E., Flandrin-Gresta, P., Nibourel, O., Preudhomme, C., Michallet, M., Thomas, X., Nicolini, F., Solly, F., Guyotat, D., Campos, L., Wattel, E., and Mortreux, F. (2016) Oncogene- and drug resistance-associated alternative exon usage in acute myeloid leukemia (AML). Oncotarget 7, 2889-2909
- Thenoz, M., Vernin, C., Mortada, H., Karam, M., Pinatel, C., Gessain, A., Webb, T. R., Auboeuf, D., Wattel, E., and Mortreux, F. (2014) HTLV-1-infected CD4+ T-cells display alternative exon usages that culminate in adult T-cell leukemia. Retrovirology 11, 119
- Vernin, C., Thenoz, M., Pinatel, C., Gessain, A., Gout, O., Delfau-Larue, M. H., Nazaret, N., Legras-Lachuer, C., Wattel, E., and Mortreux, F. (2014) HTLV-1 bZIP factor HBZ promotes cell proliferation and genetic instability by activating OncomiRs. Cancer Research 74, 6082-6093
*equal contribution
Financements :



2022-2026 ANR INFLASPLICE (ANR-21-CE15-0036-02)
2016-2020 ANR CHROTOPAS (ANR16 CE12-0009-01)
2011-2015 ANR EPIVIR
Principales collaborations
Thomas SEXTON (IGBMC, Illkirch/Strasbourg, France)
Hélène Dutartre and Chloé Journo (CIRI, Lyon, France)
Jean-Claude Twizere (GIGA, Liege, Belgium)
Anne Van den Broeke (GIGA, Liege, Belgium)
Greta Forlani and Roberto Accolla (Università degli Studi dell'Insubria - Italia)
Eric Wattel (Service d'Hématologie, Centre Hospitalier Lyon Sud, Pierre Bénite, France)
Anciens Doctorants
- Antony Ceraulo (2017-2021), MD/PhD Pediatrician HOPe, CRCL, Lyon
- Lamya Ben Ameur (2015-2019), contrat doctoral de la Ligue contre le Cancer
- Aminetou Mint Mohamed* (2012-2016), contrat doctoral de Mauritanie
- Morgan Thénoz (2010-2014), contrat de l'ecole doctorale EDISS
- Celine Vernin (2009-2013), contrat de l'ecole doctorale EDISS
- Maroun Karam (2008-2012), contrat doctoral de la FRM
- Linda Zane* (2006-2009), contrat doctoral de la Ligue contre le Cancer
- Valerie Capraro* (2005-2008), contrat de l'ecole doctorale EDISS
- Carole Pomier (2004-2008), contrat doctoral de la Ligue contre le Cancer
*co-direction
