Publication du Laboratoire de Biologie et Modélisation de la Cellule (LBMC) dans la revue Molecular Cell, le 22 août 2024. Communication du CNRS Biologie du 4 octobre 2024.
Les cassures de l’ADN sont des lésions toxiques pour la cellule. Afin de réparer ces cassures il est nécessaire, à partir de l’extrémité de ces cassures, de retrouver une séquence homologue dans l’immensité du génome. Dans un article publié dans Molecular Cell des scientifiques décrivent un modèle qui permet d’expliquer cette pêche à la séquence homologue.
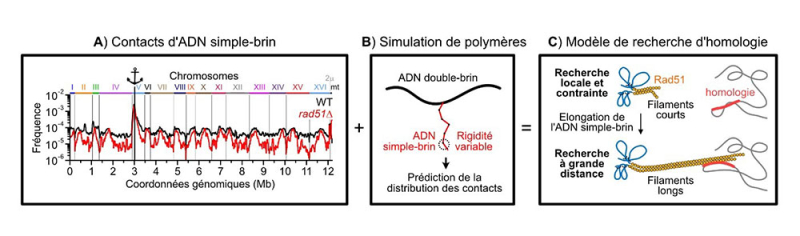
La stabilité du matériel génétique est essentielle au bon fonctionnement de chacune de nos cellules. Néanmoins des accidents peuvent se produire, par exemple sous l’effet de radiations ou d’erreurs lors de la réplication de l’ADN. Pour réparer ces cassures il existe des mécanismes extrêmement conservés chez toutes les espèces qui ont à charge d’opérer une réparation fidèle. La recombinaison homologue est une voie de réparation à haute-fidélité des cassures qui utilise une molécule d’ADN intacte de séquence identique comme matrice. Cette séquence peut provenir de la chromatide sœur, du chromosome homologue plus distant, ou d’autres séquences répétées dans le génome. Cette copie homologue intacte doit donc être identifiée dans l’immensité du génome et de l’espace nucléaire. Comme est conduite cette recherche d’une aiguille homologue dans la botte de foin du génome ?
Découvrez-en plus dans la communication réalisée par le CNRS Biologie
Référence
Mechanism of homology search expansion during recombinational DNA break repair in Saccharomyces cerevisiae. A. Dumont, N. Mendiboure, J. Savocco, L. Anani, P. Moreau, A. Thierry, L. Modolo, D. Jost, A. Piazza. Molecular Cell, 22 août 2024.
Disciplines
Mots clés






