
Publication du RDP
Vignette © Raquel Martin-Arevalillo
Les co-répresseurs sont essentiels à la régulation de la transcription chez tous les organismes vivants. Une équipe de chercheurs de Grenoble et Lyon a maintenant élucidé la structure d’un co-répresseur des plantes nommé TOPLESS. Ce travail montre que cette protéine est composée de domaines identiques à ceux d’autres co-répresseurs mais avec des propriétés différentes, illustrant comment l’évolution récupère des domaines protéiques pour créer de nouvelles fonctions. Cette étude, qui ouvre également la voie à des manipulations raisonnées de cette protéine clé pour l’adaptation des plantes à l’environnement, a été publiée le 11 juillet 2017 dans la revue PNAS.
Essentiels à la régulation de la transcription chez tous les organismes vivants, les co-répresseurs permettent de faire un lien physique entre les facteurs de transcription s’accrochant sur les promoteurs et des protéines de remodelage de la chromatine qui vont jouer sur la structure de la chromatine pour réprimer la lecture des gènes cible par la machinerie transcriptionnelle. TOPLESS est le membre fondateur d’une petite famille de co-répresseurs unique aux plantes, identifiée originellement chez la plante modèle Arabidopsis, et impliquée dans de nombreuses voies de signalisation majeurs pour le développement des plantes et leur réponse aux stress. TOPLESS interagit avec ses partenaires du fait de la présence chez ces protéines d’un motif particulier appelé le motif EAR. Son inactivation chez Arabidopsis induit la formation d’une plante où la tige est remplacée par une racine, générant ainsi une plante avec des pattes sur pattes !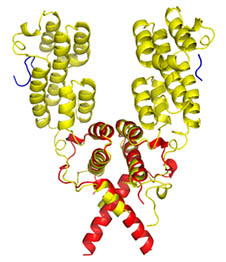
Les co-répresseurs de plante TOPLESS (jaune) et animal TBL1 (rouge) utilisent tous les deux un domaine identique (domaine LisH, partie superposée) mais de manière différente. Alors que le domaine LisH de TBL1 est impliqué dans l’interaction directe avec d’autres répresseurs, celui de TOPLESS est connecté à un second domaine (partie non superposée) responsable de l’interaction avec les répresseurs (bleu). © Renaud Dumas
Une équipe interdisciplinaire composée de généticiens moléculaires, biochimistes et biologistes structuraux du laboratoire RDP à l’ENS de Lyon (Univ. Lyon, CNRS, INRA, ENS de Lyon, UCBL) et du LPCV (Univ. Grenoble Alpes, CNRS, CEA, INRA) ainsi que de l’ESRF et de l’EMBL à Grenoble a maintenant résolu et caractérisé fonctionnellement la structure de la partie de la protéine TOPLESS d’Arabidopsis permettant l’interaction avec ses partenaires protéiques. Ce travail mené par Teva Vernoux (RDP, Lyon), Renaud Dumas (LPCV, Grenoble) et Max Nanao (ESRF et EMBL, Grenoble) a permis de montrer une structure similaire à celle du riz et conservée jusque chez les algues. La haute résolution de la structure a permis de positionner très précisément le site d’interaction avec le motif EAR de ses partenaires et l’équipe a montré que cette capacité d’interaction est également maintenue pour une protéine TOPLESS isolée chez une algue, démontrant ainsi l’ancienneté de cette fonction primordiale. La comparaison de la structure de TOPLESS avec des co-répresseurs animaux a de plus permis de mettre en évidence que ces différents co-répresseurs sont construits par assemblage de domaines identiques mais de façon différentes. Cette étude fournit ainsi un bel exemple de ce que le Prix Nobel François Jacob avait appelé le bricolage de l’évolution dans la construction de la fonction des protéines. L’étude fournit également des clés pour une manipulation de cette fonction essentielle pour les réponses aux stress environnementaux et pour améliorer le potentiel adaptatif des plantes cultivées. Cela pourrait permettre aux plantes d’éviter de finir la tête à l’envers à cause d’un stress !
En savoir plus sur le site de l'institut des sciences biologiques du CNRS
Références : Structure of the Arabidopsis TOPLESS corepressor provides insight into the evolution of transcriptional repression, Raquel Martin-Arevalillo, Max H. Nanaob, Antoine Larrieu, Thomas Vinos-Poyo, David Masta, Carlos Galvan-Ampudiad, Géraldine Brunoud, Teva Vernouxd, Renaud Dumasa and François Parcy. PNAS. doi: 10.1073/pnas.1703054114
Disciplines
Mots clés






