Communication du CNRS en date du 18 avril 2025. À partir d'une publication scientifique de Raquel Martin-Arevalillo, Bruno Guillotin, Jonas Schön, Alice Hugues, Marie-France Gerentes, Kun Tang, Jérémy Lucas, Emmanuel Thévenon, Marianne Dreuillet, Graeme Vissers, Mohammed Mohammed Ateequr, Carlos S. Galvan-Ampudia, Guillaume Cerutti, Jonathan Legrand, Coralie Cance, Annick Dubois, François Parcy, Kenneth D. Birnbaum, Matias D. Zurbriggen, Renaud Dumas, François Roudier et Teva Vernoux dans Cell.
L’auxine, une hormone végétale, agit comme un signal qui joue le chef d’orchestre pour contrôler l’expression des gènes au cours du développement des plantes. Dans un article publié dans Cell, des scientifiques ont pu décrypter le code cellulaire permettant à l’auxine de contrôler cette expression des gènes. Ils montrent que ce code repose sur une interaction combinatoire entre les nombreuses protéines qui médient la réponse à l’auxine et les différents motifs de l’ADN sur lesquels elles se lient.
L’Auxine, une molécule signal indispensable au développement de la plante
Les organismes multicellulaires, qu’ils soient végétaux ou animaux, utilisent un petit nombre de molécules qui, telles des signaux, peuvent réguler leur développement et assurer la construction de l’organisme. Comprendre comment ces signaux développementaux agissent sur le génome pour réguler l’expression des gènes dans l’espace et le temps est fondamental. Un même signal peut activer différents gènes dans différentes parties de l’organisme, mais l’orchestration de ces mécanismes reste encore énigmatique.
L’auxine, hormone essentielle impliquée dans toutes les phases de développement des plantes, est un parfait exemple de la manière dont un même signal peut être utilisé pour générer une multiplicité de profils d’expression de gènes au cours du développement.

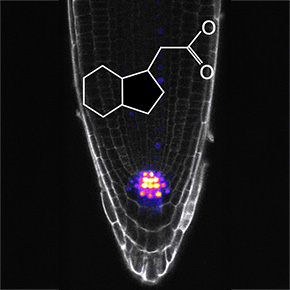
© Raquel Martin-Arevalillo et Teva Vernoux
Découvrez-en plus sur le site du CNRS
Référence
Synthetic deconvolution of an auxin-dependent transcriptional code. Raquel Martin-Arevalillo, Bruno Guillotin, Jonas Schön, Alice Hugues, Marie-France Gerentes, Kun Tang, Jérémy Lucas, Emmanuel Thévenon, Marianne Dreuillet, Graeme Vissers, Mohammed Mohammed Ateequr, Carlos S. Galvan-Ampudia, Guillaume Cerutti, Jonathan Legrand, Coralie Cance, Annick Dubois, François Parcy, Kenneth D. Birnbaum, Matias D. Zurbriggen, Renaud Dumas, François Roudier and Teva Vernoux.
Cell, 15 avril 2025, DOI : 10.1016/j.cell.2025.03.028
Disciplines
Mots clés






